Quantum ESPRESSOのpw.xで水の第一原理MDを複数回実行し、その複数のデータをDeePMD-kitで学習させ、そのDeep Potentialを使用してLAMMPSでMD計算をやってみます。
学習データの準備
PythonのASEを使って水のユニットセルを作成します。分子数は27個、密度は1 g/cm3です。
from ase import Atoms
from ase.visualize import view
from ase.build import molecule
from ase.units import mol
import numpy as np
water = molecule("H2O")
unitcell_size = 3.11
unitcell = Atoms(symbols=water.get_chemical_symbols(), positions=water.get_positions(),
cell=np.eye(3) * unitcell_size, pbc=True)
mdcell = unitcell * [3, 3, 3]
mdcell.write("water.pwi", format='espresso-in')
density = (np.sum(unitcell.get_masses())/mol) / (unitcell.get_volume()*1e-24)
print(density, "[g/cm3]")
view(mdcell)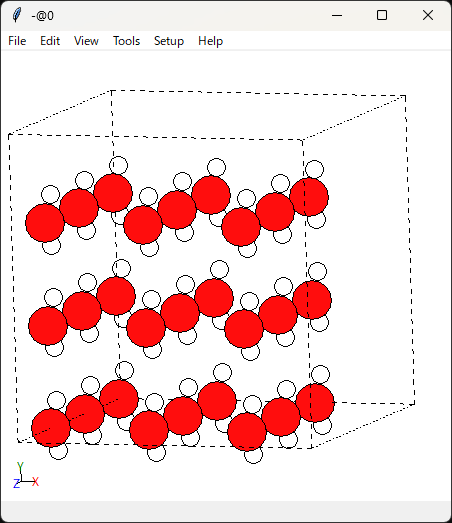
これで作成したwater.pwiを編集します。赤字のところが追記部分です。
&CONTROL
calculation='vc-md'
dt=40
nstep=500
/
&SYSTEM
ntyp = 2
nat = 81
ibrav = 0
nosym=.true.
/
&ELECTRONS
/
&IONS
ion_temperature='rescaling'
tempw=300.0
/
&CELL
cell_dynamics='pr'
press=0.001
/
ATOMIC_SPECIES
O 15.999 O.pbe-n-kjpaw_psl.0.1.UPF
H 1.008 H.pbe-rrkjus_psl.1.0.0.UPF
K_POINTS gamma
CELL_PARAMETERS angstrom
9.33000000000000 0.00000000000000 0.00000000000000
0.00000000000000 9.33000000000000 0.00000000000000
0.00000000000000 0.00000000000000 9.33000000000000
ATOMIC_POSITIONS angstrom
O 0.0000000000 0.0000000000 0.1192620000
H 0.0000000000 0.7632390000 -0.4770470000
H 0.0000000000 -0.7632390000 -0.4770470000
:
:温度300K、常圧でのNPTアンサンブルです。また、同じファイルを別フォルダにもコピーして以下の変更を行います。
- calculation=’md’
- &CELLのセクションを削除
セルサイズ変化のないMD、つまりNVTアンサンブルの計算もやります。2通りの第一原理MD計算を行い、幅広い学習データを効率よく増やす作戦です。第一原理MD計算が終わったら、トラジェクトリをASE形式に変換します。ASEを使ってDeePMD-kitの学習データを作る方法はこちらで解説しています。
DeePMD-kitのデータ作成
2つの第一原理MD計算の結果をASE形式に変換し、water_1.trajとwater_2.trajを作ります。それを以下のPythonコードでdpdata形式に変換します。
import dpdata
data = dpdata.MultiSystems.from_file(file_name="./water_1.traj", fmt="ase/structure")
data_training, data_validation, dict= data.train_test_split(40)
data_training.to_deepmd_npy('training_data_1')
data_validation.to_deepmd_npy('validation_data_1')同様にwater_2.trajをtraining_data_2, validation_data_2にします。
次に、この2つのデータを学習・検証データとするinput.jsonを作ります。
{
"_comment": " model parameters",
"model": {
"type_map": ["H", "O"],
"descriptor" :{
"type": "se_e2_a",
"sel": [70, 36],
"rcut_smth": 0.50,
"rcut": 6.00,
"neuron": [10, 20, 40],
"resnet_dt": false,
"axis_neuron": 4,
"seed": 1,
"_comment": " that's all"
},
"fitting_net" : {
"neuron": [100, 100, 100],
"resnet_dt": true,
"seed": 1,
"_comment": " that's all"
},
"_comment": " that's all"
},
"learning_rate" :{
"type": "exp",
"decay_steps": 5000,
"start_lr": 0.001,
"stop_lr": 3.51e-8,
"_comment": "that's all"
},
"loss" :{
"type": "ener",
"start_pref_e": 0.02,
"limit_pref_e": 1,
"start_pref_f": 1000,
"limit_pref_f": 1,
"start_pref_v": 0,
"limit_pref_v": 0,
"_comment": " that's all"
},
"training" : {
"training_data": {
"systems": ["./training_data_1/H54O27",
"./training_data_2/H54O27"],
"batch_size": "auto",
"_comment": "that's all"
},
"validation_data":{
"systems": ["./validation_data_1/H54O27",
"./validation_data_2/H54O27"],
"batch_size": "auto",
"numb_btch": 1,
"_comment": "that's all"
},
"numb_steps": 1000000,
"seed": 10,
"disp_file": "lcurve.out",
"disp_freq": 1000,
"save_freq": 10000,
"_comment": "that's all"
},
"_comment": "that's all"
}
内容は公式チュートリアルがベースになっています。一つ一つの設定項目についてはよく理解していませんので、不適切な設定が含まれているかもしれません。
training_dataとvalidation_dataが2つずつあるので、赤字部分で2つのパスを指定しています。この部分はPythonのリストで、文字列(データのパス)を格納する形になっています。
この時点でフォルダ構成は以下のようになります。
- input.json
- training_data_1 (training_data_2, validation_data_1, validation_data_2も同様)
- H54O27
- type.raw
- type_map.raw
- set.000
- box.npy, coord.npy, energy.npy, force.npy, virial.npy
- H54O27
ではDeep learningを開始します。以下のコマンドを実行します。
#CPUを使う場合
dp train input.json
#GPUを使う場合
CUDA_VISIBLE_DEVICES=0 horovodrun -np 1 dp train --mpi-log=workers input.json動き出すと以下のようにtrainingとvalidationのデータが2つずつ認識されて、処理が始まります。
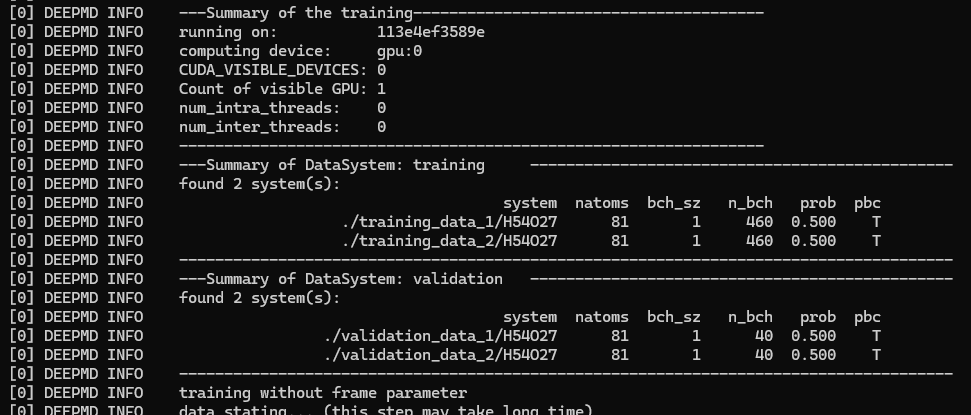
数時間後に処理が終わりました。その後は以下のコマンドでポテンシャルファイルを作成します。
dp freeze -o graph.pb
dp compress -i graph.pb -o graph-compress.pb続きはPart.2で。


コメント
training_dataとvalidation_dataが2つずつある場合のやり方が非常に参考になりました。
ありがとうございます。
続編楽しみにしております!
・QEのGPU用コンパイル
・Fe.outからFe.trajへの変換
の2つがまだ解決できなくてひたすらチャレンジしています。