Open Force Field (OpenFF)をLAMMPSで使ってみました。導入方法とLAMMPSでの使い方を解説します。
インストール方法
Windowsで使う場合はWSLの中にMinicondaを入れてそこで実行してください。
conda install mamba -c conda-forge
mamba create -n openff-toolkit -c conda-forge openff-toolkitこのコマンドでは、openff-toolkitという名前の新しいconda仮想環境が作られます。また依存関係でAmbertoolsがインストールされますが、Windows版のcondaではAmbertoolsがインストールできずエラーになります。(condaのリポジトリにはAmbertoolsのWindows版がないようです)
LinuxかMacを使うか、Windowsの場合はWSLで動かすようにしましょう。
$ conda env list
# conda environments:
#
base * /home/wsl/miniconda3
openff-toolkit /home/wsl/miniconda3/envs/openff-toolkitconda仮想環境をopenff-toolkitに切り替えます。
conda activate openff-toolkit以上で準備は完了です。
LAMMPSのデータ作成
試しにエタノール100分子のMD計算セルを作ってみます。初期密度は余裕をもって0.2 g/cm3として、さらにOpenMMによるMinimize計算をしてからLAMMPSデータを作成します。
from openff.toolkit import ForceField, Molecule, Topology
from openff.units import unit
from openff.interchange import Interchange
from openff.interchange.components._packmol import UNIT_CUBE, pack_box
from openff.interchange.components.mdconfig import MDConfig
#セルの初期密度(ボックスサイズに反映される)
density = 0.2
#中に入れる分子のSMILESと個数のリスト
# [ ["分子1", 100], ["分子2", 50] ] のような指定も可能
components = [ ["CCO", 100] ]
#Minimizeをやる場合はTrue
minimization = True
#出力するファイル名
name_in = "auto_generated.in"
name_data = "out.lmp"
molecules = []
number_of_components = []
for component in components:
molecules.append(Molecule.from_smiles(component[0]))
number_of_components.append(component[1])
topology = pack_box(
molecules=molecules,
number_of_copies=number_of_components,
mass_density=density * 1e3 * unit.kilogram / unit.meter**3,
box_shape=UNIT_CUBE,
)
print("Box vector :", topology.box_vectors)
print("Number of molecules :", topology.n_molecules)
force_field = ForceField("openff_unconstrained-2.2.0.offxml")
interchange = Interchange.from_smirnoff(force_field=force_field, topology=topology)
if minimization:
interchange.minimize()
interchange.to_lammps(name_data)
mdconfig = MDConfig.from_interchange(interchange)
mdconfig.write_lammps_input(input_file=name_in, interchange=interchange)
print("LAMMPS data file :", name_data)
print("LAMMPS in file :", name_in)これで out.lmp と auto_generated.in の2つのファイルが作成されます。ポテンシャルパラメータと座標データはout.lmpに書かれており、ポテンシャルパラメータは以下のようになりました。
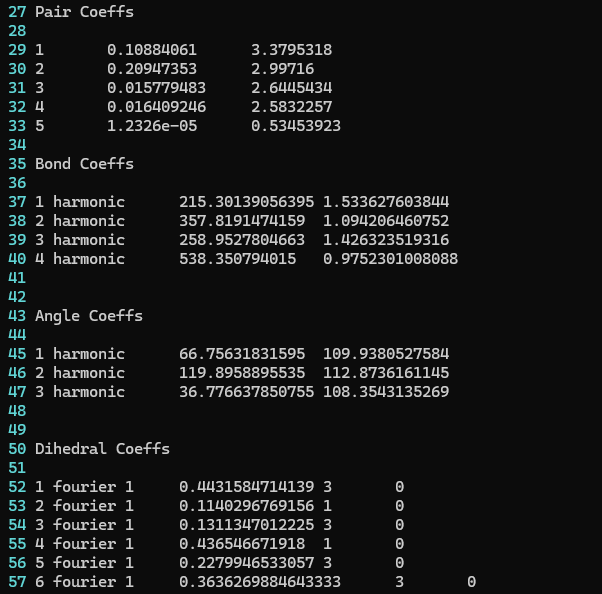
パラメータの良し悪しは現時点ではわかりませんが、それらしい値が入っています。今回はopenff_unconstrained-2.2.0.offxmlを使ってみましたが、他との違いはよく分かりません。ここで指定できるforce_fieldの一覧はこの記事の最下部に載せております。
なお、nglviewをインストールすれば topology.visualize() をすることで構造を可視化できます。
#nglviewのインストール
conda activate openff-toolkit
conda install -c conda-forge nglview
#可視化のためにJupyter Notebookを起動
jupyter notebook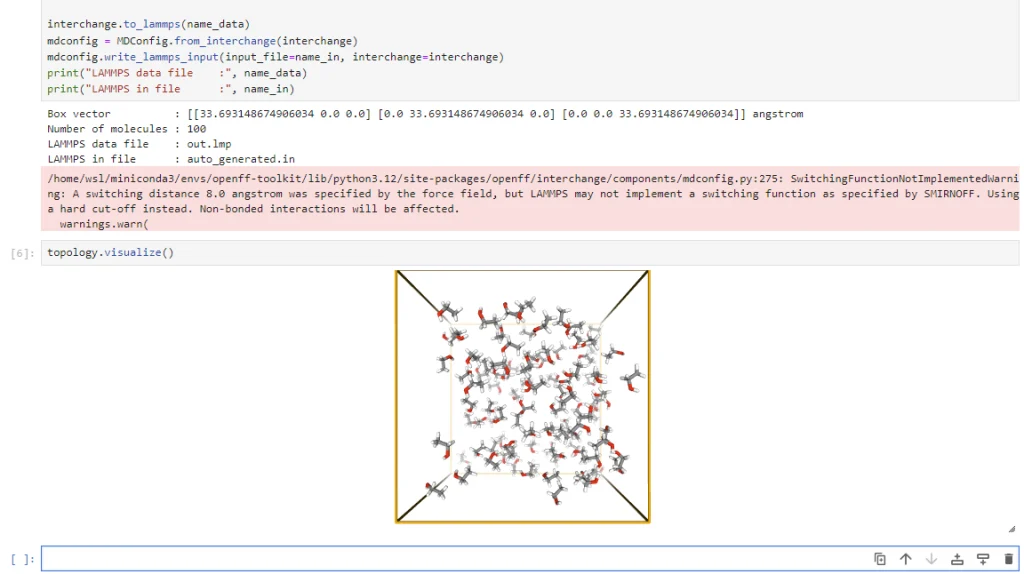
LAMMPSでMD計算
上記のコードで作成したLAMMPSデータにはDihedralの関数にfourierというものを使用しています。これを計算するためには、LAMMPSビルド時に指定するパッケージのEXTRA-MOLECULEを有効にする必要があります。それが有効になっているかどうかを確認するには、
lmp -h |grep fourierを実行して表示されたらある、なければない。無い場合はビルドをやり直します。cmakeの場合は以下のような感じです。
cmake -C ../cmake/presets/oneapi.cmake -D PKG_KSPACE=yes -D PKG_MOLECULE=yes -D PKG_RIGID=yes -D PKG_MANYBODY=yes -D PKG_OPENMP=yes -D PKG_GPU=yes \
-D PKG_EXTRA-MOLECULE=yes ../cmakeDihedralのfourierが動くようになったら、inファイルを書き換えて計算します。inファイルはとりあえず以下のようにしました。
units real
atom_style full
dimension 3
boundary p p p
bond_style hybrid harmonic
angle_style hybrid harmonic
dihedral_style hybrid fourier
special_bonds lj 0.0 0.0 0.5 coul 0.0 0.0 0.8333333333
pair_style lj/cut/coul/long 9.0 9.0
pair_modify mix arithmetic tail yes
read_data out.lmp
kspace_style ewald 1e-4
timestep 1.0
velocity all create 298.0 4928459
fix integrate all npt temp 298.0 298.0 100.0 iso 1.0 1.0 1000.0
thermo_style custom step temp press etotal pe ke density
thermo 1000
dump myDump all atom 1000 dump.lammpstrj
dump_modify myDump sort id
run 100000そして計算結果のdensityをプロットしてみます。
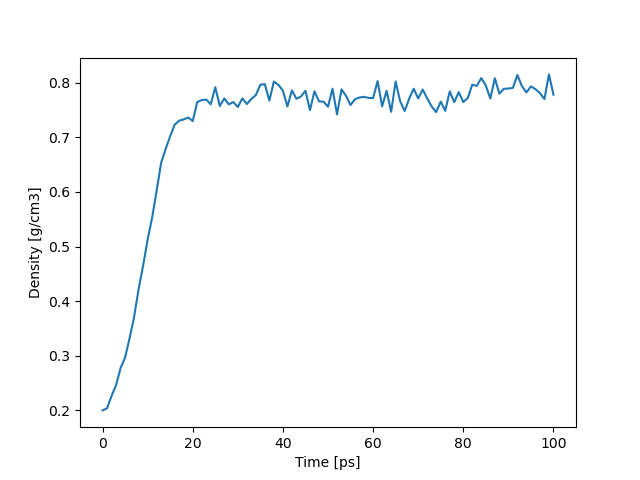
いい感じです。後半部分で平均をとると 0.7782 g/cm3で、エタノールの密度である0.789 g/cm3と非常に近いです。またトラジェクトリをASEで可視化するとこうなります。
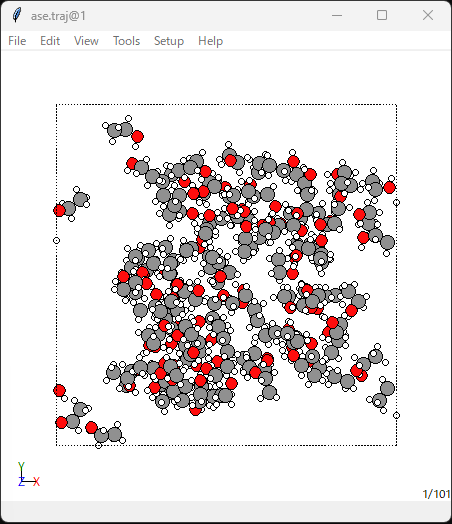
使い慣れているのでASEで可視化しましたが、OpenFFではmdtrajを使うのが一般的なようです。mdtrajはそのうち使ってみたいと思います。
ASEでの可視化方法はこちらで解説しています。
まとめ
OpenFFを使ってLAMMPSで計算を実行してみました。LAMMPSで有機分子のモデリングとポテンシャル割り当てをやろうと思うとなかなか難しいのですが、OpenFFを使うことでそれらを簡単に行うことができました。また割り当てたポテンシャルパラメータも精度が良さそうです。他にも様々な計算をOpenFFを使って試してみたいと思っていますので、結果が出たら紹介したいと思います。
補足 force_fieldsの一覧
Interchange.from_smirnoff(force_field=force_field, topology=topology) の部分で指定できるforce_fieldの一覧はこちらで確認できました。
>>> import openff.toolkit
>>> openff.toolkit.get_available_force_fields()
['ff14sb_off_impropers_0.0.4.offxml',
'ff14sb_0.0.2.offxml',
'ff14sb_0.0.4.offxml',
'ff14sb_0.0.1.offxml',
'ff14sb_0.0.3.offxml',
'ff14sb_off_impropers_0.0.2.offxml',
'ff14sb_off_impropers_0.0.1.offxml',
'ff14sb_off_impropers_0.0.3.offxml',
'smirnoff99Frosst-1.0.4.offxml',
'smirnoff99Frosst-1.0.1.offxml',
'smirnoff99Frosst-1.0.5.offxml',
'smirnoff99Frosst-1.0.9.offxml',
'smirnoff99Frosst-1.0.3.offxml',
'smirnoff99Frosst-1.0.6.offxml',
'smirnoff99Frosst-1.0.2.offxml',
'smirnoff99Frosst-1.0.0.offxml',
'smirnoff99Frosst-1.0.7.offxml',
'smirnoff99Frosst-1.0.8.offxml',
'smirnoff99Frosst-1.1.0.offxml',
'opc-1.0.1.offxml',
'openff-2.1.1.offxml',
'tip4p_ew-1.0.0.offxml',
'openff_unconstrained-1.0.0-RC2.offxml',
'openff-2.0.0.offxml',
'opc-1.0.0.offxml',
'openff-2.2.0.offxml',
'spce.offxml',
'openff-1.2.1.offxml',
'opc3-1.0.1.offxml',
'opc-1.0.2.offxml',
'openff-1.0.1.offxml',
'openff_unconstrained-1.3.1.offxml',
'openff-1.0.0-RC2.offxml',
'openff-2.0.0-rc.2.offxml',
'openff_unconstrained-2.1.1.offxml',
'spce-1.0.0.offxml',
'openff-1.3.1.offxml',
'openff_unconstrained-2.2.0-rc1.offxml',
'tip4p_fb-1.0.0.offxml',
'tip4p_ew.offxml',
'openff_unconstrained-1.0.1.offxml',
'tip3p_fb-1.0.0.offxml',
'openff-1.1.1.offxml',
'opc3.offxml',
'openff_unconstrained-2.1.0.offxml',
'openff-1.0.0-RC1.offxml',
'openff-1.3.1-alpha.1.offxml',
'tip3p_fb-1.1.1.offxml',
'openff-1.2.0.offxml',
'openff_unconstrained-1.0.0-RC1.offxml',
'openff-2.0.0-rc.1.offxml',
'opc.offxml',
'openff_unconstrained-2.1.0-rc.1.offxml',
'openff_unconstrained-2.0.0-rc.2.offxml',
'tip3p_fb.offxml',
'openff-1.0.0.offxml',
'tip3p-1.0.0.offxml',
'tip5p-1.0.0.offxml',
'openff_unconstrained-1.1.1.offxml',
'openff-2.2.0-rc1.offxml',
'openff-2.1.0-rc.1.offxml',
'tip3p_fb-1.1.0.offxml',
'openff-1.1.0.offxml',
'tip3p.offxml',
'opc3-1.0.0.offxml',
'tip4p_fb-1.0.1.offxml',
'tip5p.offxml',
'openff_unconstrained-1.2.1.offxml',
'openff_unconstrained-1.0.0.offxml',
'openff-1.3.0.offxml',
'openff_unconstrained-1.3.0.offxml',
'openff_unconstrained-2.2.0.offxml',
'openff_unconstrained-2.0.0-rc.1.offxml',
'openff_unconstrained-1.2.0.offxml',
'openff_unconstrained-2.0.0.offxml',
'openff_unconstrained-1.1.0.offxml',
'openff_unconstrained-1.3.1-alpha.1.offxml',
'tip4p_fb.offxml',
'openff-2.1.0.offxml',
'tip3p-1.0.1.offxml']

コメント